리보솜 RNA
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
리보솜 RNA(rRNA)는 리보솜을 구성하는 RNA로, mRNA 번역에 중요한 역할을 한다. rRNA는 일차 구조와 줄기-고리 구조의 이차 구조를 가지며, 리보솜 단백질과 상호작용하여 삼차 구조를 형성한다. 원핵생물과 진핵생물은 rRNA의 크기와 구성에 차이가 있으며, 16S rRNA는 계통 발생 연구에 널리 사용된다. rRNA는 리보자임으로 작용하여 펩티딜 전이 효소 중심에서 펩타이드 결합을 촉매하며, mRNA와 tRNA 결합에 영향을 미치도록 구조가 변경될 수 있다. rRNA는 rRNA의 접힘, 변형, 처리 및 리보솜 단백질과의 조립을 통해 생합성되며, 진핵생물에서는 핵소체에서, 원핵생물에서는 세포질에서 일어난다. rRNA의 생성은 세포의 성장 속도와 에너지 상태에 따라 조절되며, 비기능성 rRNA는 다양한 경로를 통해 분해된다. rRNA는 유전자 전달과 돌연변이에 대한 저항성이 강하며, 질병의 진단 및 치료, 분류학 연구에 활용된다. 인간 유전자에는 45S rRNA 전구체, 5S rRNA, 미토콘드리아 rRNA를 암호화하는 유전자가 존재한다.
더 읽어볼만한 페이지
- 리보자임 - 리보솜
리보솜은 rRNA와 단백질로 구성된 두 개의 소단위체로 이루어진 세포 내 단백질 합성 복합체로서, 세포질 내에서 mRNA의 코돈을 해독하여 폴리펩타이드 사슬을 합성하는 번역 과정을 수행하며, 원핵세포와 진핵세포의 구조적 차이로 인해 항생제 개발의 표적이 되고, RNA 세계에서 기원한 자기 복제 복합체로 추정된다. - 비부호화 RNA - CRISPR
CRISPR은 세균과 고세균이 외래 유전 물질로부터 자신을 방어하는 적응 면역 시스템으로, CRISPR-Cas9 시스템은 유전자 편집 기술로 발전하여 노벨 화학상을 수상했으며, 유전 질환 치료, 농업 등 다양한 분야에 활용될 잠재력을 지닌다. - 비부호화 RNA - 짧은 간섭 RNA
짧은 간섭 RNA(siRNA)는 RNA 간섭 경로에서 특정 유전자 발현을 억제하는 21~23 염기쌍의 짧은 이중 가닥 RNA로, RNA 유도 침묵 복합체(RISC)를 형성하여 유전자 침묵을 유도하며, 생의학 연구 및 신약 개발 분야에서 주목받고 있으나, 표적 외 효과 및 면역계 유발 가능성 등의 과제도 존재한다.
2. 구조
리보솜 RNA(rRNA)는 염기 서열의 일차 구조와 염기쌍 간 상호작용으로 형성되는 줄기-고리 구조(2차 구조)를 가진다. 종에 따라 줄기-고리의 길이와 위치는 다르지만, 3차원 구조는 유사하다.[4] rRNA는 리보솜 단백질과 특이적 상호작용을 통해 리보솜 소단위를 구성하는데, 리보솜 단백질은 염기성/방향족 아미노산 잔기(페닐알라닌, 티로신, 트립토판)를 통해 rRNA와 스태킹 상호 작용을 하거나, 염기성 잔기(라이신, 아르기닌)를 가진 결합 부위로 rRNA의 당-인산 골격과 가교 결합한다. 이러한 상호작용과 소형 및 대형 리보솜 소단위 결합으로 단백질 합성이 가능한 기능적 리보솜이 생성된다.[5]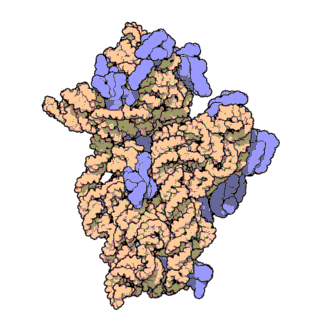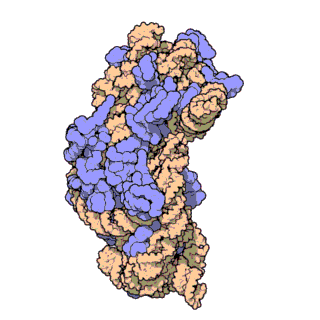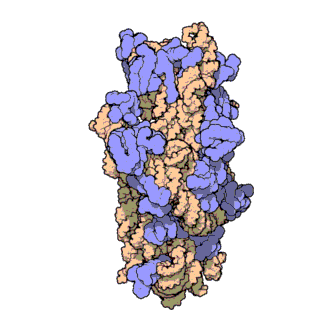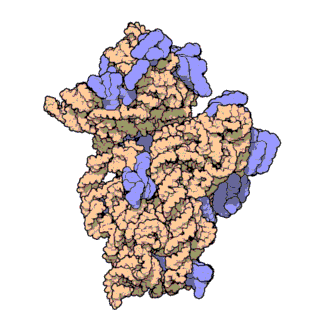
rRNA는 대형 소단위(LSU)와 소형 소단위(SSU)로 구성되며, 함께 작용하여 기능하는 리보솜을 형성한다. 원핵생물과 진핵생물에서 크기와 구성에 차이가 있다.
- 원핵생물: LSU는 50S, SSU는 30S이다. SSU는 단일 소형 rRNA 분자(~1500 뉴클레오티드), LSU는 단일 소형 rRNA와 단일 대형 rRNA 분자(~3000 뉴클레오티드)를 포함하며, 23S, 5S rRNA(LSU)와 16S rRNA(SSU)의 세 가지 유형이 ~50개의 리보솜 단백질과 결합한다.
- 진핵생물: LSU는 60S, SSU는 40S이다. SSU는 단일 소형 rRNA(~1800 뉴클레오티드), LSU는 2개의 소형 rRNA와 1개의 대형 rRNA 분자(~5000 뉴클레오티드)를 포함하며, 70개 이상의 리보솜 단백질과 상호작용하여 더 크고 다형성인 리보솜 단위를 형성한다.[6] LSU에 3종, SSU에 1종으로 총 4가지 유형의 rRNA가 있다.[7]
효모는 진핵생물 rRNA 연구 모델이었으나, 최근 극저온 전자 현미경 발전으로 다른 진핵생물 리보솜 연구도 가능해졌다.[8] 효모 LSU는 5S, 5.8S, 28S rRNA를 포함하며, 5.8S와 28S는 팽창 세그먼트(ES)가 없는 원핵생물 23S rRNA와 크기/기능이 유사하다. 최근 아스가르드 고세균(Lokiarchaeota, Heimdallarchaeota) 23S rRNA에서 두 개의 초대형 ES가 발견되어 진핵생물 rRNA와 연관성이 주목받고 있다.[9] 염호성 고세균 ''Halococcus morrhuae'' 5S rRNA에는 108 뉴클레오티드 삽입이 있다.[10][11] 진핵생물 SSU는 ES를 포함하는 18S rRNA 소단위를 가진다.
SSU 및 LSU rRNA 서열은 계통 발생 관계 연구에 널리 사용된다. 고대 기원이며,[12] 모든 생명체에서 발견되고 수평 유전자 전달에 저항하며, 리보솜 기능에 중요하여 시간이 지나도 보존되기 때문이다.[13] 16S rRNA는 원핵생물 종 구분에 주요하게 사용된다.[14]
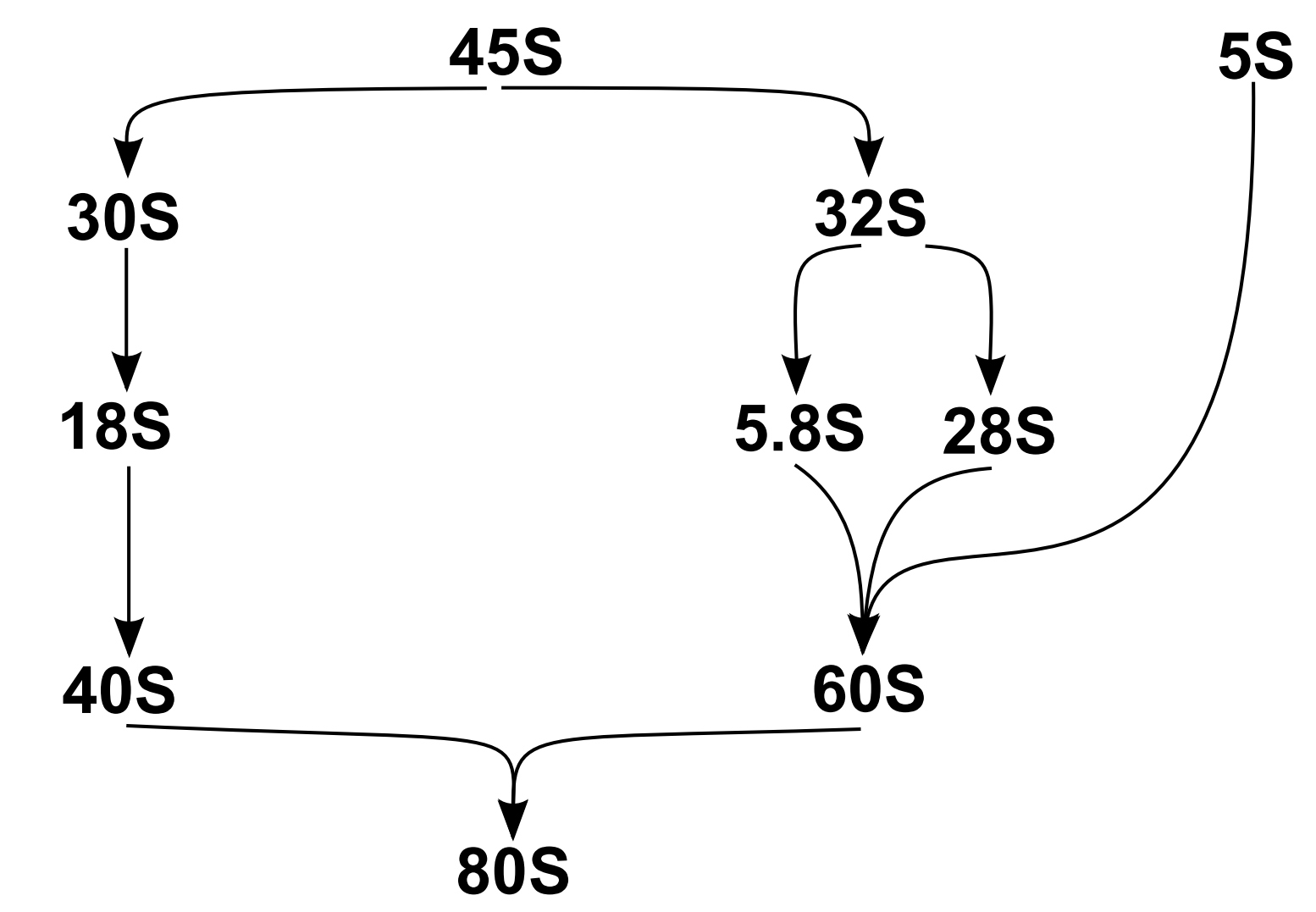
원핵생물과 진핵생물 리보솜은 아래 표와 같이 두 개의 소단위체로 나눌 수 있다.
소단위체의 S 단위는 침강 속도 측정값이므로 단순 합산할 수 없다. nt 단위는 rRNA 중합체의 선형 단위 정수이므로 더할 수 있다.
rRNA를 암호화하는 유전자 클러스터는 rDNA라고 불린다. rRNA는 유전자 전달, 돌연변이 등에 대한 저항성이 있어 생물 연구에 중요하며,[62] 유전자 데이터베이스 프로젝트(SILVA[63], SINA[64] 등)에서 리보솜 RNA 서열 정렬은 분류 할당, 계통 발생 분석 등에 활용된다.[63]
2. 1. 리보자임
대형 소단위(LSU) rRNA 아형은 펩티딜 전이 효소 중심(PTC)에서 리보솜 단백질이 리보솜의 촉매 부위에 결합할 수 없기 때문에 리보자임이라고 불린다.[15]소형 소단위(SSU) rRNA 아형은 해독 중심(DC)에서 mRNA를 해독한다.[16] 리보솜 단백질은 DC에 들어갈 수 없다.
2. 2. 형태 변화
rRNA의 구조는 다른 mRNA의 번역 과정에서 리보솜에 대한 tRNA 결합에 영향을 미치기 위해 극적으로 변경될 수 있다.[17] 16S rRNA에서 이는 rRNA의 특정 뉴클레오타이드가 한 뉴클레오타이드 또는 다른 뉴클레오타이드 사이에서 염기쌍을 교환하여 rRNA의 형태를 변경하는 "스위치"를 형성할 때 발생하는 것으로 생각된다. 이 과정은 LSU와 SSU의 구조에 영향을 미쳐, rRNA 구조의 이러한 형태 변화 스위치가 tRNA 선택에서 코돈을 안티코돈과 일치시키고 mRNA를 해독하는 능력에 있어 전체 리보솜에 영향을 미친다는 것을 시사한다.[18]3. 기능
rRNA는 mRNA 번역의 촉매 부위를 형성하는데 중요한 역할을 한다. mRNA와 tRNA를 결합하여 mRNA의 코돈 서열을 아미노산으로 번역하는 과정을 촉진한다. rRNA는 tRNA가 소형 소단위(SSU)와 대형 소단위(LSU) 사이에 끼어 있을 때 단백질 합성을 촉매한다. SSU에서 mRNA는 tRNA의 안티코돈과 상호작용하고, LSU에서 tRNA의 아미노산 수용체 줄기는 LSU rRNA와 상호작용한다. 리보솜은 에스테르-아미드 교환을 촉매하여, 생성된 펩타이드의 C-말단을 tRNA에서 아미노산의 아민으로 전달한다.[15]
리보솜에는 A, P, E 세 개의 결합 부위가 있다.
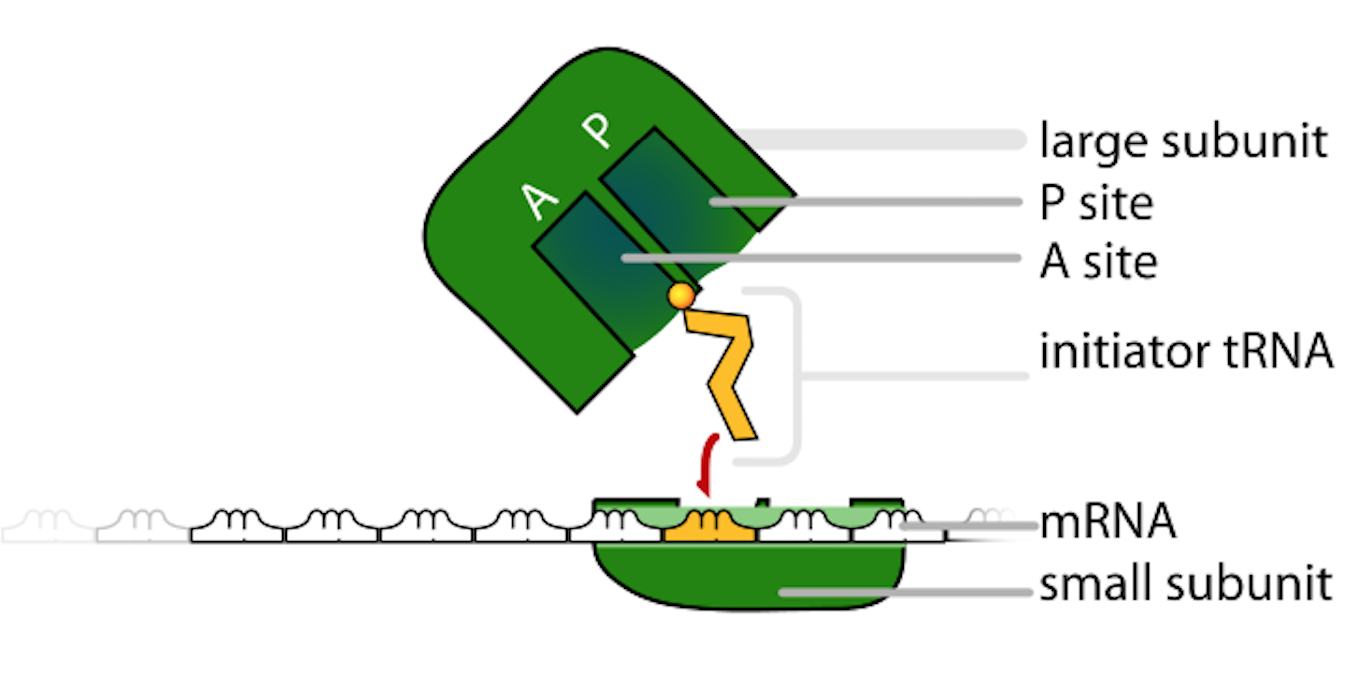
하나의 mRNA는 여러 개의 리보솜에 의해 동시에 번역될 수 있는데, 이를 폴리솜이라고 한다.
원핵생물에서 A 부위는 주로 16S rRNA로 구성되어 있다. P 부위에서는 결정 구조를 통해 16s rRNA의 3' 말단이 mRNA 분자처럼 부위로 접힐 수 있다는 것이 밝혀졌다. 이는 소단위체를 안정화하는 분자 간 상호 작용을 유발한다. 펩티딜 전이 효소 중심은 23S rRNA 소단위체의 뉴클레오타이드에 의해 형성된다.[15] E 부위에는 더 많은 단백질이 포함되어 있으며, tRNA가 16S 및 23S rRNA 소단위체와 모두 결합하는 것으로 나타났다.[21]
4. 생합성
리보솜 RNA (rRNA)의 합성과 리보솜으로의 조립은 rRNA의 접힘, 변형, 처리 및 리보솜 단백질과의 조립을 통해 대형 리보솜 소단위체(LSU)와 소형 리보솜 소단위체(SSU)를 형성하는 과정이다. 원핵생물은 막으로 둘러싸인 세포 소기관이 없기 때문에 rRNA 합성은 세포질에서 일어난다. 반면 진핵생물의 경우 이 과정은 주로 핵소체에서 일어나며, pre-RNA 합성이 먼저 시작된다. RNA 중합효소 I에 의한 pre-RNA 전사는 세포 전체 RNA 전사의 약 60%를 차지한다.[19] pre-RNA는 엔도뉴클레아제와 엑소뉴클레아제, RNA 헬리케이스, GTPase 및 ATPase에 의해 촉매되는 과정을 통해 접히고 리보솜 단백질과 조립된다. rRNA는 외부 전사 스페이서와 내부 전사 스페이서를 제거하기 위해 엔도뉴클레아제 및 엑소뉴클레아제에 의해 처리된다.[20] pre-RNA는 리보솜 조립 인자 및 리보솜 단백질과 조립되어 pre-리보솜 입자를 형성하기 전, 메틸화 또는 유사유리딘화와 같은 변형을 거친다. 이후 핵소체에서 세포질로 이동하여 성숙 단계를 거친 후, 이 입자들은 결합하여 리보솜을 형성한다.[20] rRNA의 1차 구조 내 염기성 및 방향족성 잔기는 스태킹 상호작용과 리보솜 단백질에 대한 인력을 통해 rRNA 골격과 리보솜 단위의 다른 구성 요소 사이에 가교 효과를 만든다.
진핵생물은 일반적으로 직렬 반복되는 rRNA 유전자의 많은 사본을 가지고 있다. 인간의 경우, 약 300~400개의 반복이 5개의 클러스터에 존재하며, 염색체 13 (RNR1), 14 (RNR2), 15 (RNR3), 21 (RNR4), 22 (RNR5)에 위치한다. 이배체 인간은 총 rDNA 10개의 클러스터를 가지며, 이는 전체 인간 게놈의 0.5% 미만이다.[29]
포유류 세포는 2개의 미토콘드리아 rRNA (12S, 16S) 분자와 4가지 유형의 세포질 rRNA (28S, 5.8S, 18S, 5S 소단위체)를 가진다. 28S, 5.8S, 18S rRNA는 2개의 내부 전사 스페이서로 분리된 단일 전사 단위 (45S)에 의해 암호화된다.[28] 45S rDNA는 염색체 13, 14, 15, 21, 22에 5개의 클러스터 (각각 30–40개 반복)로 구성되며, RNA 중합효소 I에 의해 전사된다. 5S 소단위체 DNA는 직렬 배열 (~200–300개의 실제 5S 유전자와 많은 분산된 유사 유전자)로 발생하며, 가장 큰 것은 염색체 1q41-42에 있다. 5S rRNA는 RNA 중합효소 III에 의해 전사된다. 대부분의 진핵생물에서 18S rRNA는 작은 리보솜 소단위체에, 큰 소단위체에는 3개의 rRNA 종 (포유류의 5S, 5.8S, 28S, 식물의 25S rRNA)이 포함된다.
파리에서 큰 소단위체는 5.8S rRNA가 더 짧은 5.8S 소단위체 (123 nt)와 2S rRNA (30 뉴클레오티드)로 분할되어 4개의 rRNA 종을 포함한다. 두 단편은 28 뉴클레오티드의 내부 전사 스페이서로 분리된다. 2S rRNA는 작고 매우 풍부하여 sRNA 라이브러리 구성에 간섭하고 다른 sRNA의 정량화를 손상시킬 수 있다. 2S 소단위체는 초파리 및 검은날개 버섯파리 종에서 발견되지만 모기에서는 발견되지 않는다.[32]
작은 소단위체 리보솜 RNA (SSU rRNA)의 3차 구조는 X선 결정학으로 밝혀졌다.[33] SSU rRNA의 2차 구조는 5', 중앙, 3' 주요, 3' 부 도메인의 4개 도메인을 포함한다.
rRNA의 생산은 세포 소기관인 리보솜 합성의 속도 제한 단계이다.
4. 1. 진핵생물에서의 생합성
RNA 중합효소 I은 핵소체에서 rDNA를 전사하여 18S, 28S, 5.8S rRNA를 포함하는 pre-rRNA 분자를 생성한다. rRNA 합성은 리보솜 합성의 속도 제한 단계이다. pre-rRNA는 외부 및 내부 스페이서 서열에 의해 분리된 후 메틸화되어 조립 및 단백질 접힘에 중요한 역할을 한다.[19] 5S rRNA 유전자는 핵소체 내부에 위치하며 RNA 중합효소 III에 의해 pre-5S rRNA로 전사된다. pre-5S rRNA는 LSU를 형성하기 위해 28S 및 5.8S rRNA와 함께 처리 및 조립을 위해 핵소체로 들어간다.[20]4. 2. 원핵생물에서의 생합성
원핵생물에서 rRNA 생성은 리보솜 합성의 속도 제한 단계이다. ''대장균''에서 rRNA는 7개의 서로 다른 ''rrn'' 오페론 내에 있는 두 개의 프로모터 P1과 P2로부터 전사된다.[51][52][53] P1 프로모터는 중간에서 높은 박테리아 성장 속도 동안 rRNA 합성을 조절하는 데 특히 중요하다. 이 프로모터의 전사 활성은 성장 속도에 정비례하므로 rRNA 조절에 주로 관여한다. rRNA 농도가 증가하면 리보솜 합성에 대한 음성 피드백 메커니즘 역할을 한다. 높은 NTP 농도는 ''rrn'' P1 프로모터의 효율적인 전사에 필요한 것으로 밝혀졌다. 이들은 RNA 중합 효소 및 프로모터와 안정적인 복합체를 형성하는 것으로 여겨진다. 특히 세균에서 높은 NTP 농도와 rRNA 합성이 증가하는 이러한 연관성은 리보솜(단백질 합성)이 성장 속도에 의존하는 이유에 대한 분자적 설명을 제공한다. 낮은 성장 속도는 더 낮은 rRNA/리보솜 합성 속도를, 더 높은 성장 속도는 더 높은 rRNA/리보솜 합성 속도를 나타낸다. 이를 통해 세포는 필요와 사용 가능한 자원에 따라 에너지를 절약하거나 대사 활동을 증가시킬 수 있다.[51][52][53]원핵 세포에서 각 rRNA 유전자 또는 오페론은 16S, 23S, 5S rRNA 및 tRNA 서열과 전사된 스페이서를 포함하는 단일 RNA 전구체로 전사된다. RNA 가공은 전사가 완료되기 전에 시작된다. 가공 반응 동안 rRNA와 tRNA는 별도의 분자로 방출된다.[54]
5. 조절
리보솜 RNA(rRNA)의 합성은 여러 단계의 조절 과정을 거쳐 이루어진다.
진핵생물에서 리보솜 RNA 합성은 다양한 과정과 상호작용을 통해 항상성을 유지하기 위해 상향 조절 및 하향 조절된다. 키나아제의 일종인 AKT는 RNA 중합효소 I의 활성에 영향을 주어 간접적으로 rRNA 합성을 촉진한다.[46] 특정 혈관 신생 리보뉴클레아제인 안지오제닌(ANG)은 핵으로 이동하여 핵소체에 축적될 수 있는데, ANG의 농도가 지나치게 높아지면 프로모터 영역의 rDNA에 결합하여 rRNA 전사를 불필요하게 증가시킬 수 있으며, 이는 핵소체에 해롭고, 무제한 전사와 암으로 이어질 수 있다.[47] 세포 내 포도당이 제한적인 상황에서는 AMP 활성화 단백질 키나아제(AMPK)가 에너지를 소비하지만 필수적이지 않은 대사 과정을 억제하는데, 결과적으로 AMPK는 RNA 중합효소 I을 인산화(Ser-635 부위에서)하여 전사 개시를 방해함으로써 rRNA 합성을 하향 조절한다.[48] 리보솜 해독 센터에서 하나 이상의 슈도유리딘 또는 2'-O-메틸화 영역이 손상되거나 제거되면 새로운 아미노산의 통합 속도가 감소하여 rRNA 전사 속도가 크게 감소한다.[49] 헤테로크로마틴 형성은 rRNA 전사를 억제하는 데 필수적인데, 이 과정이 없으면 리보솜 RNA가 무제한으로 합성되어 생물의 수명이 크게 감소한다.[50]
원핵생물에서 rRNA 생성은 리보솜 합성의 속도 제한 단계이다. 대장균에서 rRNA는 7개의 ''rrn'' 오페론 내 두 개의 프로모터 P1과 P2로부터 전사된다. P1 프로모터는 성장 속도에 비례하여 rRNA 합성을 조절하며, rRNA 농도가 증가하면 리보솜 합성에 대한 음성 피드백 메커니즘으로 작용한다. 높은 NTP 농도는 ''rrn'' P1 프로모터의 효율적인 전사에 필요하며, 이는 리보솜 합성이 성장 속도에 의존하는 이유를 설명한다.[51][52][53] rRNA 조절 메커니즘은 여러 중복을 가진다. 전사 수준에서 긍정적 및 부정적 효과자가 존재하여 세포 항상성 유지를 돕는다. ''rrn'' P1 프로모터 상류의 UP 요소는 RNA 중합 효소 서브유닛과 결합하여 rRNA 전사를 촉진하고, FIS와 같은 전사 인자는 프로모터 상류에 결합하여 RNA 중합 효소와 상호작용하여 전사를 촉진한다. 종결 방지 인자는 ''rrn'' P2 프로모터 하류에 결합하여 조기 전사 종결을 방지하며, 엄격한 반응으로 인해, 아미노산 가용성이 낮을 때 ppGpp(부정적 효과자)는 P1 및 P2 프로모터 모두에서 전사를 억제한다.[51]
5. 1. 진핵생물에서의 조절
리보솜 RNA의 합성은 다양한 과정과 상호 작용을 통해 항상성을 유지하기 위해 상향 조절 및 하향 조절된다.- 키나아제 AKT는 간접적으로 rRNA 합성을 촉진하는데, 이는 RNA 중합효소 I이 AKT에 의존하기 때문이다.[46]
- 특정 혈관 신생 리보뉴클레아제인 안지오제닌(ANG)은 핵으로 이동하여 핵소체에 축적될 수 있다. ANG의 농도가 너무 높아지면, 일부 연구에 따르면 ANG는 프로모터 영역의 rDNA에 결합하여 불필요하게 rRNA 전사를 증가시킬 수 있다. 이는 핵소체에 해로울 수 있으며, 무제한 전사와 암으로 이어질 수도 있다.[47]
- 세포 내 포도당 제한 상황에서 AMP 활성화 단백질 키나아제(AMPK)는 에너지를 소비하지만 필수적이지 않은 대사 과정을 억제한다. 결과적으로, AMPK는 RNA 중합효소 I을 인산화(Ser-635 부위에서)하여 전사 개시를 방해함으로써 rRNA 합성을 하향 조절할 수 있다.[48]
- 리보솜 해독 센터에서 하나 이상의 슈도유리딘 또는 2'-O-메틸화 영역의 손상 또는 제거는 새로운 아미노산의 통합 속도를 감소시켜 rRNA 전사 속도를 상당히 감소시킨다.[49]
- 헤테로크로마틴의 형성은 rRNA 전사를 침묵시키는 데 필수적이며, 이 과정이 없으면 리보솜 RNA가 무제한으로 합성되어 유기체의 수명이 크게 감소한다.[50]
5. 2. 원핵생물에서의 조절
원핵생물에서 리보솜 RNA(rRNA) 생성은 리보솜 합성의 속도 제한 단계이다. 대장균에서 rRNA는 7개의 ''rrn'' 오페론 내 두 개의 프로모터 P1과 P2로부터 전사된다. P1 프로모터는 성장 속도에 비례하여 rRNA 합성을 조절하며, rRNA 농도가 증가하면 리보솜 합성에 대한 음성 피드백 메커니즘으로 작용한다. 높은 NTP 농도는 ''rrn'' P1 프로모터의 효율적인 전사에 필요하며, 이는 리보솜 합성이 성장 속도에 의존하는 이유를 설명한다.[51][52][53]rRNA 조절 메커니즘은 여러 중복을 가진다. 전사 수준에서 긍정적 및 부정적 효과자가 존재하여 세포 항상성 유지를 돕는다.
6. 분해
리보솜 RNA는 다른 일반적인 유형의 RNA에 비해 상당히 안정적이며 건강한 세포 환경에서 더 오랫동안 지속된다. 일단 기능 단위로 조립되면, 리보솜 내의 리보솜 RNA는 세포 주기 정지기 동안 수 시간 동안 안정적이다.[55] 리보솜 RNA의 분해는 리보솜이 결함이 있는 mRNA를 인식하거나, 번역을 중단시키는 다른 처리 문제에 직면하여 "정지"될 때 유발될 수 있다. 리보솜이 정지되면, 특화된 경로를 통해 전체 복합체의 분해가 시작된다.[56]
6. 1. 진핵생물에서의 분해
모든 단백질 또는 RNA와 마찬가지로, rRNA 생성은 오류가 발생하기 쉬우며, 이는 기능하지 않는 rRNA의 생성을 초래한다. 이를 수정하기 위해 세포는 비기능성 rRNA 분해(NRD) 경로를 통해 rRNA의 분해를 허용한다.[57] 이 주제에 대한 대부분의 연구는 진핵 세포, 특히 ''사카로마이세스 세레비지애'' 효모에서 수행되었다. 현재, 세포가 기능적으로 결함이 있는 리보솜을 유비퀴틴화 및 분해를 위해 어떻게 표적화하는지에 대한 기본적인 이해만이 진핵생물에서 가능하다.[58]- 40S 소단위에 대한 NRD 경로는 60S 소단위에 대한 NRD 경로와 독립적이거나 분리될 수 있다. 특정 유전자가 특정 pre-RNA의 분해에는 영향을 미치지만 다른 pre-RNA에는 영향을 미치지 못하는 것이 관찰되었다.[59]
- Mms1p 및 Rtt101p와 같은 수많은 단백질이 NRD 경로에 관여하며, 이들은 함께 복합체를 이루어 리보솜을 분해 대상으로 삼는 것으로 여겨진다. Mms1p와 Rtt101p가 함께 결합하는 것으로 밝혀졌으며, Rtt101p는 유비퀴틴 E3 리가아제 복합체를 모집하여, 기능하지 않는 리보솜이 분해되기 전에 유비퀴틴화되도록 하는 것으로 여겨진다.[60]
- 진핵 세포의 성장률은 비기능성 rRNA의 축적에 의해 크게 영향을 받지 않는 것으로 보였다.
6. 2. 원핵생물에서의 분해
원핵생물의 리보솜 RNA 분해에 대한 연구는 진핵생물에 비해 훨씬 적지만, 세균이 진핵생물의 NRD와 유사한 분해 방식을 따르는지에 대한 관심은 여전히 존재해 왔다. 원핵생물에 대한 연구는 대부분 ''대장균''에서 수행되었다. 진핵생물과 원핵생물의 rRNA 분해 사이에는 많은 차이점이 발견되었으며, 연구자들은 두 생물이 다른 경로를 사용하여 분해된다고 믿게 되었다.[61]7. 서열 보존성과 안정성
리보솜 RNA(rRNA)는 모든 생명체에 널리 존재하며, 그 서열은 시간이 지나도 크게 변하지 않는 특성을 지니고 있다.[12] 이러한 안정성 덕분에 rRNA는 유전자 전달, 돌연변이, 생물 파괴 없이 일어나는 변화를 연구하는 데 매우 중요하다.[13] rRNA 유전자는 변형과 침입에 강한 것으로 알려져 있으며, rRNA 서열 변화는 세포 기능에 심각한 손상을 초래할 수 있다.
rRNA 서열은 리보솜의 기능에 매우 중요한 역할을 하기 때문에 오랜 시간이 지나도 잘 보존된다.[13] 특히 16S rRNA 계통 분석 정보는 비슷한 원핵생물 종을 구분하는 주요 방법으로 사용된다.[14]
SILVA, SINA와 같은 유전자 데이터베이스 프로젝트는 리보솜 RNA 서열을 정렬하여 생물의 분류, 계통 발생 분석, 미생물 다양성 조사를 돕는다.
8. 의의
rRNA는 모든 세포에 존재하는 몇 안 되는 유전자 산물 중 하나이다.[45] rRNA를 암호화하는 유전자(rDNA)는 유기체의 분류학적 그룹 식별, 관련 그룹 계산, 종 발산 속도 추정에 사용된다.[68] RDP-II[69] 및 SILVA[70]와 같은 특수 데이터베이스에는 수천 개의 rRNA 염기서열이 저장되어 있다.
rRNA의 변화는 ''결핵균''(결핵 유발 세균)과 같은 특정 질병 유발 세균의 약물 내성을 유발할 수 있다.[71] rRNA는 클로람페니콜, 에리스로마이신 등 여러 임상 관련 항생제의 표적이다. 또한, rRNA는 miR-663, miR-712와 같은 종 특이적 마이크로 RNA의 기원이기도 하다.[73]
이러한 rRNA의 특성은 진화뿐만 아니라 분류학, 의학 분야에서도 중요하다. 특히 rRNA 연구는 질병 진단 및 치료 기술 개발에 활용될 수 있다.
9. 인간 유전자
10. 관련 용어
- 16S rRNA 계통 분석
- rDNA
참조
[1]
서적
Molekulare Zellbiologie
DE GRUYTER
1996-01-31
[2]
웹사이트
Molecular Expressions Cell Biology: Ribosomes
https://micro.magnet[...]
National High Magnetic Field Laboratory
2015-11-13
[3]
웹사이트
Ribosome Definition, Function, Formation, Role, Importance, & Facts Britannica
https://www.britanni[...]
2024-03-08
[4]
논문
The Three Roles of RNA in Protein Synthesis
https://www.ncbi.nlm[...]
2000
[5]
논문
Protein-rRNA binding features and their structural and functional implications in ribosomes as determined by cross-linking studies
1995-09
[6]
논문
Roles of eukaryotic ribosomal proteins in maturation and transport of pre-18S rRNA and ribosome function
2005-10
[7]
논문
5 S rRNA: structure and interactions
2003-05
[8]
논문
An overview of pre-ribosomal RNA processing in eukaryotes
2015-03-01
[9]
논문
Supersized ribosomal RNA expansion segments in Asgard archaea
2020-08
[10]
논문
An archaebacterial 5S rRNA contains a long insertion sequence.
1981-05
[11]
논문
Cryo-electron microscopy visualization of a large insertion in the 5S ribosomal RNA of the extremely halophilic archaeon ''Halococcus morrhuae''
2020-08-31
[12]
논문
Phylogenetic structure of the prokaryotic domain: the primary kingdoms
1977-11
[13]
논문
RNAmmer: consistent and rapid annotation of ribosomal RNA genes
2007-05-01
[14]
논문
EzTaxon: a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences
2007-10
[15]
논문
The Peptidyl Transferase Center: a Window to the Past
2021-11
[16]
논문
Eukaryote-specific extensions in ribosomal proteins of the small subunit: Structure and function
2015-01-02
[17]
논문
A conformational switch in Escherichia coli 16S ribosomal RNA during decoding of messenger RNA
1997-08
[18]
논문
Major rearrangements in the 70S ribosomal 3D structure caused by a conformational switch in 16S ribosomal RNA
1999-11
[19]
논문
Ribosome biogenesis in the yeast Saccharomyces cerevisiae
2013-11
[20]
논문
Eukaryotic Ribosome Assembly
2019-06
[21]
논문
The involvement of RNA in ribosome function
2002-07
[22]
논문
Homo sapiens RNA, 5S ribosomal
http://www.ncbi.nlm.[...]
2020-09-03
[23]
논문
''Homo sapiens'' 5.8S ribosomal RNA
https://www.ncbi.nlm[...]
2017-02-10
[24]
논문
''Homo sapiens'' 28S ribosomal RNA
https://www.ncbi.nlm[...]
2017-02-04
[25]
논문
''Homo sapiens'' 18S ribosomal RNA
https://www.ncbi.nlm[...]
2017-02-04
[26]
논문
The 55S mammalian mitochondrial ribosome and its tRNA-exit region.
2015-07
[27]
논문
rrnDB: improved tools for interpreting rRNA gene abundance in bacteria and archaea and a new foundation for future development
2015-01
[28]
논문
The function and synthesis of ribosomes
2001-07
[29]
논문
Human rRNA gene clusters are recombinational hotspots in cancer
2009-12
[30]
논문
Variation in human chromosome 21 ribosomal RNA genes characterized by TAR cloning and long-read sequencing
2018-07
[31]
논문
Variant ribosomal RNA alleles are conserved and exhibit tissue-specific expression
2018-02
[32]
논문
Distribution of split 5.8S ribosomal RNA in Diptera
http://dx.doi.org/10[...]
1992-08
[33]
논문
Crystal structure of the ribosome at 5.5 A resolution
2001-05
[34]
웹사이트
Ribosomal RNA genetics
https://www.britanni[...]
[35]
논문
RNA folding in living cells
2010-11
[36]
논문
Crystal structure of the 14-subunit RNA polymerase I
https://resolver.sub[...]
2013-10-01
[37]
논문
RNA polymerase I structure and transcription regulation
2013-10-01
[38]
논문
The initial U3 snoRNA:pre-rRNA base pairing interaction required for pre-18S rRNA folding revealed by in vivo chemical probing
2011-07-01
[39]
논문
RNA folding pathways and the self-assembly of ribosomes
2011-12-01
[40]
논문
Tuning the ribosome: The influence of rRNA modification on eukaryotic ribosome biogenesis and function
2017-09-01
[41]
논문
A cluster of methylations in the domain IV of 25S rRNA is required for ribosome stability
2014-10-01
[42]
논문
Methylation of 12S rRNA is necessary for in vivo stability of the small subunit of the mammalian mitochondrial ribosome
2009-04-01
[43]
논문
Nucleolar clustering of dispersed tRNA genes
2003-11-01
[44]
웹사이트
rRNA synthesis and processing
https://www.nobelpri[...]
[45]
논문
Evolutionary rates vary among rRNA structural elements
[46]
논문
AKT promotes rRNA synthesis and cooperates with c-MYC to stimulate ribosome biogenesis in cancer
2011-08-01
[47]
논문
Angiogenin as a molecular target for the treatment of prostate cancer
2011-05-01
[48]
논문
AMP-activated protein kinase adapts rRNA synthesis to cellular energy supply
2009-10-01
[49]
논문
Loss of rRNA modifications in the decoding center of the ribosome impairs translation and strongly delays pre-rRNA processing
2009-09-01
[50]
논문
Heterochromatin formation promotes longevity and represses ribosomal RNA synthesis
2012-01-01
[51]
논문
Transcription regulation by initiating NTP concentration: rRNA synthesis in bacteria
1997-12-01
[52]
논문
Strength and Regulation of Seven rRNA Promoters in Escherichia coli
2015-12-30
[53]
논문
Colocalization of distant chromosomal loci in space in E. coli: a bacterial nucleolus
2016-10-01
[54]
서적
Molecular and Cellular Biology
Wadsworth Publishing Company
[55]
논문
Ribosome degradation in growing bacteria
2011-05-01
[56]
논문
Ribosome-associated protein quality control
2016-01-01
[57]
논문
A role for ubiquitin in the clearance of nonfunctional rRNAs
2009-04-01
[58]
논문
Investigating nonfunctional rRNA decay as a stress response in Saccharomyces cerevisiae
https://www.fasebj.o[...]
2011-04-01
[59]
논문
A late-acting quality control process for mature eukaryotic rRNAs
2006-11-01
[60]
논문
A role for Saccharomyces cerevisiae Cul8 ubiquitin ligase in proper anaphase progression
2003-06-01
[61]
논문
The effects of disruptions in ribosomal active sites and in intersubunit contacts on ribosomal degradation in Escherichia coli
2015-01-01
[62]
논문
Abundance of ribosomal RNA gene copies maintains genome integrity
2010-02-01
[63]
논문
The SILVA ribosomal RNA gene database project: improved data processing and web-based tools
2013-01-01
[64]
논문
SINA: accurate high-throughput multiple sequence alignment of ribosomal RNA genes
2012-07-01
[65]
논문
Aptazyme-mediated regulation of 16S ribosomal RNA
2010-03-01
[66]
논문
A genomic-library based discovery of a novel, possibly synthetic, acid-tolerance mechanism in Clostridium acetobutylicum involving non-coding RNAs and ribosomal RNA processing
2010-05-01
[67]
논문
The dormancy regulator DosR controls ribosome stability in hypoxic mycobacteria
2012-07-01
[68]
논문
Fast evolving 18S rRNA sequences from Solenogastres (Mollusca) resist standard PCR amplification and give new insights into mollusk substitution rate heterogeneity
2010-03-01
[69]
논문
The Ribosomal Database Project (RDP-II): previewing a new autoaligner that allows regular updates and the new prokaryotic taxonomy
2003-01-01
[70]
논문
SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB
[71]
논문
Tuberculosis and the Tubercle Bacillus
American Society of Microbiology
[72]
논문
Single 23S rRNA mutations at the ribosomal peptidyl transferase centre confer resistance to valnemulin and other antibiotics in Mycobacterium smegmatis by perturbation of the drug binding pocket
2009-03-00
[73]
논문
The atypical mechanosensitive microRNA-712 derived from pre-ribosomal RNA induces endothelial inflammation and atherosclerosis
[74]
서적
基礎分子生物学(第2版)
東京化学同人
[75]
논문
Comparative analysis of ribosomal proteins in complete genomes: An example of reductive evolution at the domain scale
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com

